Utilizzando l’intelligenza artificiale, DeepMind prevede la struttura di quasi tutte le proteine conosciute. Ciò consente, tra l’altro, di comprendere meglio le malattie e di sviluppare farmaci più rapidamente. “Sta rivoluzionando le scienze della vita”.
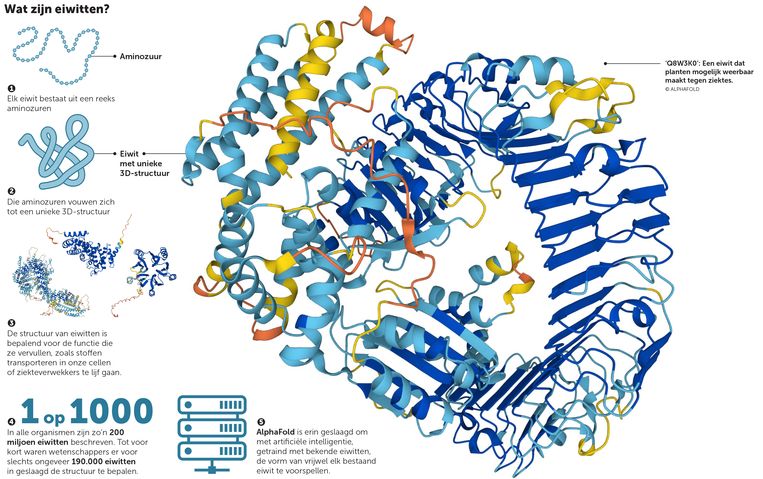
Se vuoi capire la vita sulla terra, devi capire come funzionano le proteine. Diventerà molto più facile per gli scienziati ora che la società britannica DeepMind, parte di Google dal 2014, ha caricato la struttura di circa 200 milioni di proteine utilizzando l’intelligenza artificiale.
L’algoritmo AlphaFold prevede l’aspetto di praticamente tutte le proteine conosciute. Non solo le circa 20.000 proteine del nostro stesso organismo, ma anche quelle di piante, animali e microrganismi. “Questo rivela la struttura dell’universo delle proteine”, ha affermato Demis Hassabis, CEO di DeepMind. “Siamo all’alba di una nuova era in biologia”.
“AlphaFold sta rivoluzionando le scienze della vita”, conferma il biotecnologo Lennart Martens (UGent e Istituto fiammingo di biotecnologia). “Le proteine sono gli strumenti della biologia e, quando conosci la loro forma, puoi capire meglio cosa fanno. Proprio come un martello è diverso da un cacciavite.
Il codice per produrre le proteine è memorizzato nel nostro DNA. Le proteine sono costituite da catene di amminoacidi e un gene contiene le istruzioni per legare insieme gli amminoacidi in un certo ordine. Una proteina si ripiega quindi in un’unica struttura tridimensionale. “Sappiamo che le informazioni necessarie sono memorizzate nella proteina, quindi in linea di principio è possibile prevedere la forma di una proteina in base alla sequenza di amminoacidi”, afferma il biologo molecolare Han Remaut (VUB e VIB). “Ma non siamo ancora stati in grado di comprendere appieno quelle regole”.
opera titanica
Questo rende il districare le strutture proteiche un compito arduo. “È un processo costoso e laborioso”, afferma Remaut. “Possono volerci anni per svelare la struttura di una proteina”. Gli scienziati lo fanno, tra le altre cose, irradiando proteine con raggi X e deducendo la struttura della dispersione del raggio.
Fino a poco tempo, gli scienziati erano riusciti a determinare sperimentalmente la struttura di circa 190.000 proteine. DeepMind ha addestrato il suo algoritmo con sequenze di amminoacidi e strutture proteiche note. AlphaFold cerca modelli in questa grande quantità di dati. L’algoritmo utilizza questa conoscenza per prevedere la struttura di altre proteine.
L’anno scorso AlphaFold ha già pubblicato la struttura di tutte le proteine umane e reso pubblicamente disponibile il software, in modo che i ricercatori possano lavorare con loro stessi. Da allora, il numero di studi pubblicati che utilizzano l’algoritmo è esploso. “Ha davvero accelerato la ricerca”, afferma Remaut, che utilizza anche AlphaFold stesso. Ora che quasi tutte le proteine conosciute si trovano in un database disponibile pubblicamente, la loro struttura viene determinata in base a un commento di revisione. Scienza facile come una ricerca su google.
Sviluppo farmacologico più rapido
AlphaFold è particolarmente utile nella ricerca medica e nello sviluppo di farmaci. “La struttura di una proteina ci dice perché a volte smette di funzionare e può farci ammalare”, afferma Remaut. “E le interazioni tra le proteine determinano se agenti patogeni come batteri e virus riescono a infettare i nostri corpi. Grazie a questa tecnologia, possiamo comprendere meglio questi processi.
Quasi tutti i farmaci agiscono sulle proteine. Il loro scopo è attivare o inattivare determinate proteine consentendo a un’altra proteina di legarsi ad esse. Confrontalo con il modo in cui una chiave si inserisce in una serratura. Se non sai che aspetto hanno la serratura e le possibili chiavi, devi testare migliaia di molecole in modo abbastanza casuale fino a trovare una chiave che corrisponda. “Con la conoscenza della struttura, non navighiamo più alla cieca e lo sviluppo di farmaci può essere notevolmente accelerato”, afferma Martens.
Non che tutti i problemi biologici siano stati risolti con questo. AlphaFold non prevede sempre con precisione la struttura delle proteine. “Alcune proteine non sono forti come un martello, ma flessibili come un asciugamano”, spiega Martens. “L’algoritmo sta ancora lottando con quello.” Secondo l’Istituto europeo di bioinformatica (EBI), che sta lanciando il database, circa il 35% delle strutture sono altamente accurate. Circa il 45% è meno accurato, ma sufficiente per far avanzare la ricerca.
Sembra strano che DeepMind condivida la sua esperienza in modo apparentemente disinteressato. “Non era l’intenzione originale”, dice Remaut. “L’azienda ha una fortuna con questo. Ma potrebbe essere stato deciso che non sarebbe etico non condividere questa conoscenza a causa della sua grande importanza. Inoltre, in DeepMind, vogliono utilizzare la biologia sintetica per creare molecole personalizzate. Forse l’azienda lo vede principalmente come un modello di business, e questo è solo un primo passo.
Le applicazioni non si limitano alla medicina. Ad esempio, gli scienziati britannici stanno utilizzando AlphaFold per cercare proteine in grado di scomporre la plastica e i ricercatori norvegesi stanno utilizzando la tecnologia per conoscere le proteine essenziali per il sistema immunitario delle api. “È una miniera d’oro per la scienza”, afferma Martens. “E abbiamo appena iniziato a esplorarlo.”
“Fanatico di Twitter. Piantagrane. Fanatico del bacon malvagio. Giocatore sottilmente affascinante. Esperto di birra.”
You may also like
-
Conseguenze negative per la salute se i pazienti rinunciano alle cure del medico di famiglia
-
A Phoenix la temperatura è stata di oltre 43 gradi per diciannove giorni • Il fiume scorre fino alle pareti del Taj Mahal
-
Attivi tutti i giorni o solo nei fine settimana? Entrambi fanno bene al cuore
-
Il buco nero supermassiccio della Via Lattea ha avuto una grande esplosione 200 anni fa
-
L’aspartame, il dolcificante delle bevande dietetiche, aumenta il rischio di cancro? Salute e scienza